環境: macOS Sierra 10.12, 3.3 GHz Intel Core i5, メモリ8 GB.
参考: bioinformatics > 構造予測 > MODELLER > 簡単な実行例 (2015.05.03)
PKCδ C1Bドメインの結晶構造1ptr.pdbは,LYS234, ARG273, そしてGLU274の側鎖が欠けています.また,MDシミュレーションのために,N末端とC末端にGLY残基を付加したい.以前は,Swiss-pdb viewerで簡単に出来ていましたが,macOS Sierraで起動しなかったので,PymolとModellerを使いました.
最近はホモロジーモデリングサーバーがWebに多くありますが,ローカルでもホモロジーモデリングが出来るようにMODELLER (version 9.17) をインストールして使います.
$ brew tap salilab/salilab
$ brew install modellerEdit /usr/local/Cellar/modeller/9.17/modlib/modeller/config.py
and replace XXXX with your Modeller license key
Pymolで1PTRを開き,N末端のカルボニル炭素を選択 (control+中クリック or 右ダブルクリック) してから,menu > Build > Residue > Glycineを選択してGLY残基を付加.同様に,N末端の窒素原子を選択して,GLY残基を付加します (本当のシークエンスではPROです).N末端に付加したGLY残基が周囲とぶつかっているので,手動で逃がしてあげます.
Mouse Modeを3-Button Editingに切り替えてから,control+右クリックで結合を選択します.表示されたホイールをドラッグして二面角を変化させました.File > Save Molecule > OK > 1PTR_patch.pdbとして保存.
1PTR_patch.pdbをテキストエディタで開き,亜鉛 (ZN) 原子とリガンド (PRB) の行をタンパク質の後ろに移動させて保存します.
次にModellerのためのアラインメントファイルを作成します.ホモロジーモデリングを行う時はタンパク質配列のアラインメントはClustal Omegaなどのサービスで行うことができます.今回は,側鎖の補完なので以下のようにしました.アラインメントファイル中のdot (.) はtemplate構造をコピーし剛体として扱うことを意味します.
align.pirを以下のように作ります.ホモロジーモデリングで亜鉛イオンを扱いたい時は,記号はzです.
>P1;TARGET sequence:TARGET:::::::: G...K......................................RE......G...* >P1;1PTR_patch structureX:1PTR_patch:230:A:284:A:::: G...K......................................RE......G...*
計算を実行するためのスクリプトファイルmodel.pyを作ります.
from modeller import *
from modeller.automodel import *
log.verbose()
env = environ()
# Read in HETATM records from template PDBs
env.io.hetatm = True
a = automodel(env,
alnfile = 'align.pir',
knowns = '1PTR_patch',
sequence = 'TARGET',
assess_methods=(assess.DOPE,
#soap_protein_od.Scorer(),
assess.GA341))
a.starting_model= 1
a.ending_model =10
a.make()
MODELLERを実行します.
$ mod9.17 model.py
今回はモデルを10個生成し,15秒程で計算が終わりました.model.logファイルの最後にモデルの評価値が書かれています.
>> Summary of successfully produced models: Filename molpdf DOPE score GA341 score ---------------------------------------------------------------------- TARGET.B99990001.pdb 1105.60205 -3754.83594 1.00000 TARGET.B99990002.pdb 1112.45032 -3754.93115 1.00000 TARGET.B99990003.pdb 1102.25818 -3705.38794 1.00000 TARGET.B99990004.pdb 1112.61877 -3740.67261 1.00000 TARGET.B99990005.pdb 1100.41248 -3773.57837 1.00000 TARGET.B99990006.pdb 1110.39478 -3751.47119 1.00000 TARGET.B99990007.pdb 1108.92419 -3746.39502 1.00000 TARGET.B99990008.pdb 1109.27356 -3732.12354 1.00000 TARGET.B99990009.pdb 1099.53418 -3808.25830 1.00000 TARGET.B99990010.pdb 1100.34485 -3785.94385 1.00000
molpdfもDOPE scoreも値が低い程評価が高いことを意味します.今回は9番目のモデルがどちらの評価でも最低値でした.
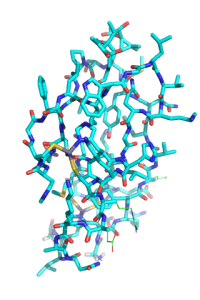 PymolでTARGET.B99990009.pdbと鋳型の1PTR_patch.pdbを開いて構造を確認します (鋳型をstick表示,モデルをline表示).LYS234, ARG273, そしてGLU274の側鎖が付加され,N末端およびC末端に付加したGLY残基もわずかに最適化されていました.
PymolでTARGET.B99990009.pdbと鋳型の1PTR_patch.pdbを開いて構造を確認します (鋳型をstick表示,モデルをline表示).LYS234, ARG273, そしてGLU274の側鎖が付加され,N末端およびC末端に付加したGLY残基もわずかに最適化されていました.
(了)