環境: macOS Sierra 10.12.5, CPU: 3.3 GHz Intel Core i5, メモリ: 8 GB. Python 3.6.1, Keras 2.0.6, TensorFlow 1.3.0rc0, RDKit 2017.03.3, mordred 0.4.0.post1.dev1.
参考サイト:
- Wash that gold- Modelling solubility with Molecular fingerprints by Esben Jannik Bjerrum
- Chainer: クラス分類による溶解度の予測 – Cheminformist3
人工知能が話題なので,見様見真似でニューラルネットワークを使った化合物の物性予測モデルの構築をやってみました.機械学習のライブラリはGoogleが開発・使用しているTensorFlowです.KerasはTensorFlowのニューラルネットワークの機能を使い易くしてくれるパッケージです(Theanoというライブラリも使える).
Contents
準備1
pyenv + pyenv-virtualenvで仮想環境を構築します (参考サイト: Pythonの環境構築 on Mac ( pyenv, virtualenv, anaconda, ipython notebook )).
$ brew install gcc open-babel
$ brew install pyenv
$ brew install pyenv-virtualenv
~/.bash_profileに以下の内容を追加.
PYENV_ROOT=~/.pyenv export PATH=$PATH:$PYENV_ROOT/bin eval "$(pyenv init -)" eval "$(pyenv virtualenv-init -)"
$ source ~/.bash_profile
最新のパッケージマネージャーをインストールします.Numpyなど科学計算に必要なパッケージは大抵デフォルトでインストールされます.
$ pyenv install --list
$ pyenv install anaconda3-4.4.0
$ pyenv global anaconda3-4.4.0
(anaconda3-4.4.0)$ conda update conda
分子記述子を扱うためのRDKitとmordredを導入します (参考サイト: mordred-descriptor).
(anaconda3-4.4.0)$ conda install -c rdkit -c mordred-descriptor mordred
rdkitのバージョンは2017.03.3,mordredのバージョンは0.4.0.post1.dev1でした.
準備2
機械学習に必要なパッケージをインストールします.
(anaconda3-4.4.0)$ pip install keras
TensorFlowは
(anaconda3-4.4.0)$ pip install tensorflowでコンパイル済のものをインストールできます.これはCPU拡張命令(AVX2など)を使用できないので,ソースからコンパイルしてインストールする方法を下に記録しておきます(参考サイト: Installing TensorFlow from Sources – TensorFlow).
(anaconda3-4.4.0)$ brew install bazel
(anaconda3-4.4.0)$ git clone https://github.com/tensorflow/tensorflow
(anaconda3-4.4.0)$ cd tensorflow
(anaconda3-4.4.0)$ git checkout r1.2
(anaconda3-4.4.0)$ ./configure
#Returnを連打
(anaconda3-4.4.0)$ bazel build --config=opt //tensorflow/tools/pip_package:build_pip_package
#時間がかかる (約25分)
(anaconda3-4.4.0)$ bazel-bin/tensorflow/tools/pip_package/build_pip_package /tmp/tensorflow_pkg
(anaconda3-4.4.0)$ pip install /tmp/tensorflow_pkg/tensorflow-1.3.0rc0-cp36-cp36m-macosx_10_7_x86_64.whl
TensorFlowの動作確認.
(anaconda3-4.4.0)$ ipython
import tensorflow as tf
hello = tf.constant('Hello, TensorFlow!')
sess = tf.Session()
print(sess.run(hello))
“b’Hello, TensorFlow!'”と表示されればOK.
jupyter-notebookを起動します (ブラウザが開く).
(anaconda3-4.4.0)$ jupyter-notebook
New▼ → Notebook: Python 3 で新しいノートブックを作成します.Shift + returnでコマンドが実行できます.終了はFile → Close and haltでノートブックを閉じた後,terminalでcontrol + Cでサーバーをshutdownします.
Neural network model with mordred and Keras
Molecular neural network models with RDKit and Keras in Pythonを参考に化合物の溶解度予測モデルを構築してみます.RDKitのレポジトリから,分子の構造と溶解度のデータが含まれるsolubility.train.sdf(1025分子)とsolubility.test.sdf(257分子)をダウンロードします.後々面倒なので,この2つのファイルを1つのファイルにします.
(anaconda3-4.4.0)$ obabel solubility.train.sdf solubility.test.sdf -O solubility.sdf
以下の作業はjupyter-notebookで行います.新規ファイルを作成したら,以下の内容をコピペして,shift-returnで実行できます.
#モジュールの読み込み from rdkit import Chem from rdkit.Chem.Draw import IPythonConsole from mordred import descriptors, Calculator import numpy as np from sklearn.preprocessing import StandardScaler from sklearn import model_selection from keras.models import Sequential from keras.layers import Dense, Activation from keras.optimizers import SGD
calc = Calculator(descriptors, ignore_3D = True)
#読み込むファイル
sdf = [ mol for mol in Chem.SDMolSupplier('solubility.sdf')]
#mordredを使ってsdfファイル中の分子の化学記述子 (原子数や分子量など分子を表現する数値) を計算.
X = calc.pandas(sdf).astype('float').dropna(axis = 1)
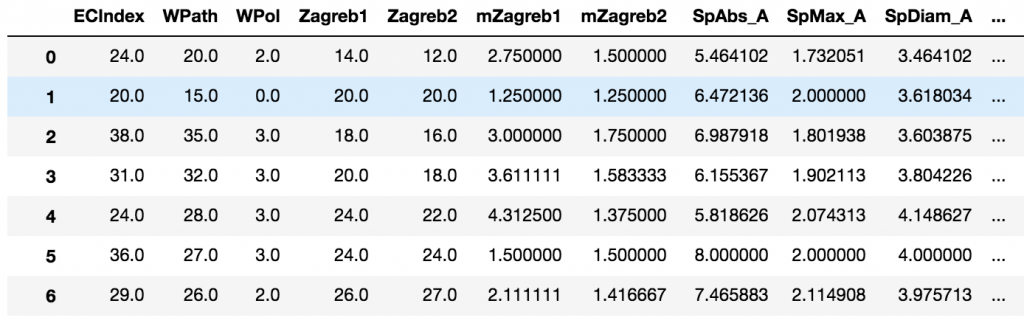
1282分子を処理するのに33秒かかりました.mordredが計算できる記述子は2次元記述子が1610種類, 3次元記述子が214種類です.今回は2次元のみ計算し,値が計算できなかった記述子をdropna(axis = 1) で削っているので,得られた記述子は1114種類でした.
試しに
X
と打って,shift + returnすると中身が表示されます.分子の数が1282,変数 (記述子) の数が1114です
#Numpy形式の配列に変換
X = np.array(X, dtype = np.float32)
#各記述子について平均0, 分散1に変換
st = StandardScaler()
X= st.fit_transform(X)
#後で再利用するためにファイルに保存
np.save("X_2d.npy", X)
溶解度をsdfファイルから読み出す (参考: Build regression model in Keras).
#溶解度を読み出す関数を定義
def getResponse( mols, prop= "SOL" ):
Y = []
for mol in mols:
act = mol.GetProp( prop )
Y.append( act )
return Y
#溶解度をsdfファイルから読み込む
Y = getResponse(sdf)
#Numpy形式の配列に変換
Y = np.array(Y, dtype = np.float32)
#後で再利用するためにファイルに保存
np.save("Y_2d.npy", Y)
#訓練セットとテストセットに再分割(ランダム).
X_train, X_test, y_train, y_test = model_selection.train_test_split(X,
Y, test_size=0.25, random_state=42)
np.save("X_train.npy", X_train)
np.save("X_test.npy", X_test)
np.save("y_train.npy", y_train)
np.save("y_test.npy", y_test)
ニューラルネットワーク層を上から順番に記述します.下の場合は単層ニューラルネットワークになります.
model = Sequential()
#入力層.Denseは全結合層の意味.次の層に渡される次元は50.入力データの次元(input_dim)は1114.
model.add(Dense(units = 50, input_dim = X.shape[1]))
model.add(Activation("sigmoid"))
#出力層.次元1,つまり一つの値を出力する.
model.add(Dense(units = 1))
model.summary()
#SGDはStochastic Gradient Descent (確率的勾配降下法).局所的最小値にとどまらないようにする方法らしい.nesterovはNesterovの加速勾配降下法.
model.compile(loss = 'mean_squared_error',
optimizer = SGD(lr = 0.01, momentum = 0.9, nesterov = True),
metrics=['accuracy'])
history = model.fit(X_train, y_train, epochs = 100, batch_size = 32,
validation_data = (X_test, y_test))
score = model.evaluate(X_test, y_test, verbose = 0)
print('Test loss:', score[0])
print('Test accuracy:', score[1])
y_pred = model.predict(X_test)
rms = (np.mean((y_test - y_pred) ** 2)) ** 0.5
#s = np.std(y_test - y_pred)
print("Neural Network RMS", rms)
計算は5秒弱で終了しました.
Epoch 499/500 961/961 [==============================] - 0s - loss: 0.1511 - acc: 0.0166 - val_loss: 0.7379 - val_acc: 0.0125 Epoch 500/500 961/961 [==============================] - 0s - loss: 0.1454 - acc: 0.0166 - val_loss: 0.7537 - val_acc: 0.0125 Test loss: 0.428150146735 Test accuracy: 0.0155763239875 Neural Network RMS 2.81453055091
結果の可視化
import matplotlib.pyplot as plt
plt.figure()
plt.scatter(y_train, model.predict(X_train), label = 'Train', c = 'blue')
plt.title('Neural Network Predictor')
plt.xlabel('Measured Solubility')
plt.ylabel('Predicted Solubility')
plt.scatter(y_test, model.predict(X_test), c = 'lightgreen', label = 'Test', alpha = 0.8)
plt.legend(loc = 4)
plt.show()
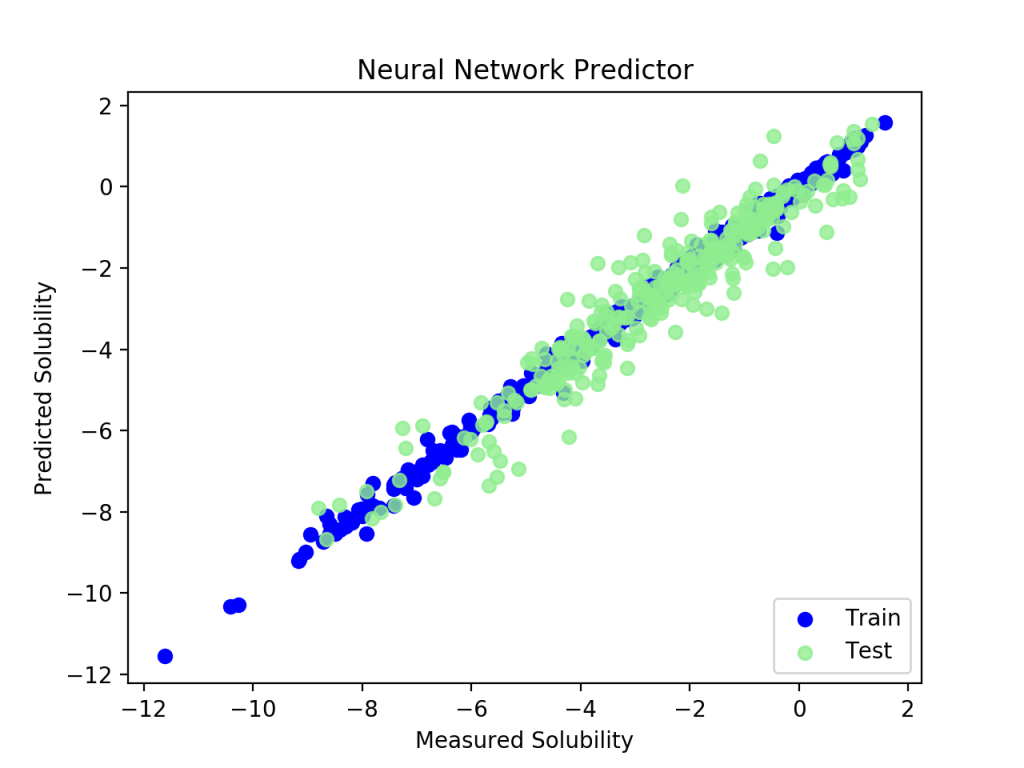
訓練セットはぴったり回帰されていますがテストセットはばらけているので,過学習に陥いっています.この程度の計算で100 epochは回し過ぎのようです.訓練セットのloss値とテストセットのloss値をプロットしてみます.
import matplotlib.pyplot as plt
loss = history.history['loss']
val_loss = history.history['val_loss']
epochs = len(loss)
plt.plot(range(epochs), loss, marker = '.', label = 'loss')
plt.plot(range(epochs), val_loss, marker = '.', label = 'val_loss')
plt.legend(loc = 'best')
plt.grid()
plt.xlabel('epoch')
plt.ylabel('loss')
plt.show()
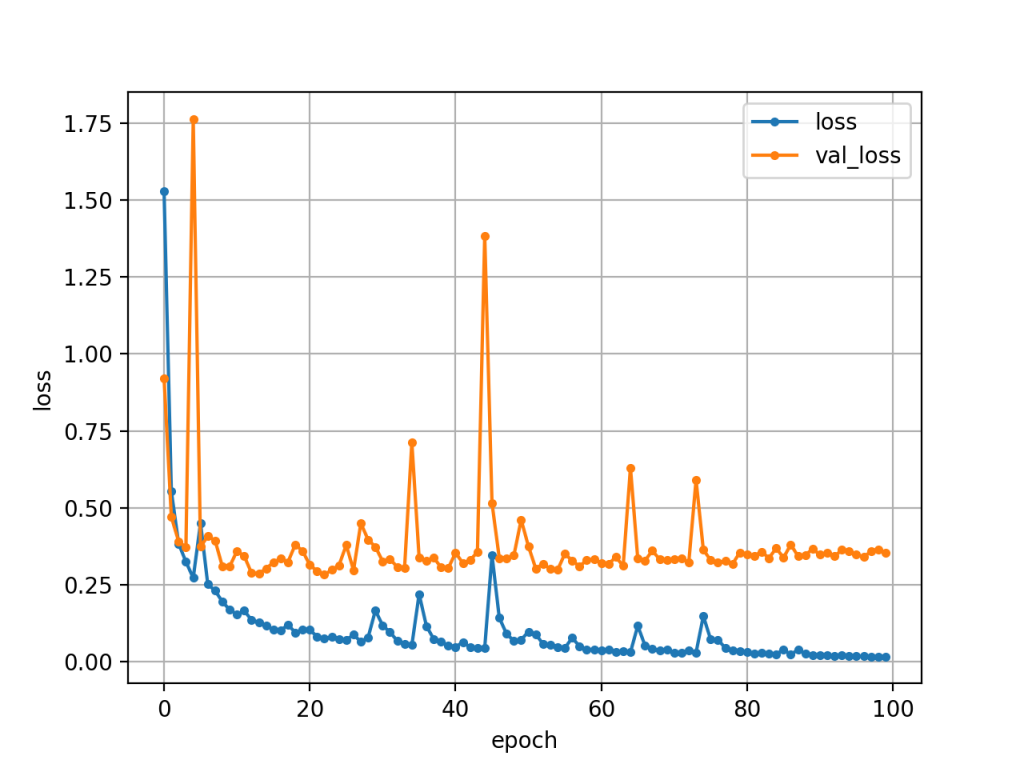
途中からテストセットのlossが漸増しています.12epochぐらいで打ち切った方がよさげです.
過学習を防止するため,val_lossに変化がなくなった段階で訓練を打ち切るEarlyStoppingが有効です.
model.compile(loss = 'mean_squared_error',
optimizer = SGD(lr = 0.01, momentum = 0.9, nesterov = True),
metrics=['accuracy'])
from keras.callbacks import EarlyStopping
history = model.fit(X_train, y_train, epochs = 100, batch_size = 32,
validation_data=(X_test, y_test), callbacks = [EarlyStopping()])
score = model.evaluate(X_test, y_test, verbose = 0)
print('Test loss:', score[0])
print('Test accuracy:', score[1])
y_pred = model.predict(X_test)
rms = (np.mean((y_test - y_pred) ** 2)) ** 0.5
#s = np.std(y_test - y_pred)
print("Neural Network RMS", rms)
Epoch 6/500 961/961 [==============================] - 0s - loss: 0.2442 - acc: 0.0104 - val_loss: 0.3072 - val_acc: 0.0156 Epoch 7/500 961/961 [==============================] - 0s - loss: 0.2215 - acc: 0.0083 - val_loss: 0.9356 - val_acc: 0.0062 Test loss: 0.935621553887 Test accuracy: 0.00623052959502 Neural Network RMS 2.86083230159
すぐに計算が打ち切られました.過学習も防ぐことができていました.
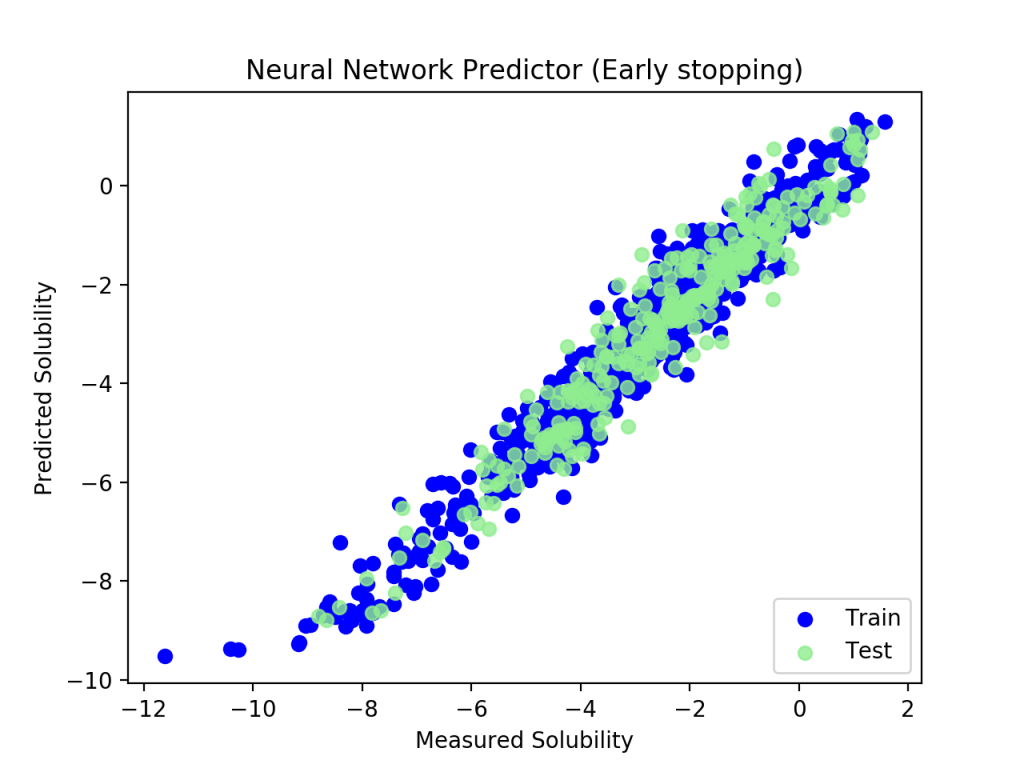
KerasRegressor
Build regression in Kerasを参考にKerasRegressorを使う.
import numpy as np
from sklearn import model_selection
from sklearn.metrics import mean_squared_error
from sklearn.metrics import r2_score
from keras.models import Sequential
from keras.layers import Activation, Dense
from keras.wrappers.scikit_learn import KerasRegressor
from keras.optimizers import SGD
#保存済みファイルを読み込む
X_train = np.load("X_train.npy")
y_train = np.load("y_train.npy")
X_test = np.load("X_test.npy")
y_test = np.load("y_test.npy")
def base_model():
model = Sequential()
model.add( Dense( units = 50, input_dim = X_train.shape[1] ) )
model.add( Activation( "sigmoid" ) )
model.add( Dense( units = 1 ) )
model.compile( loss = 'mean_squared_error', optimizer = SGD(lr = 0.01, momentum = 0.9, nesterov = True),
metrics=['accuracy'] )
return model
estimator = KerasRegressor( build_fn = base_model,
epochs = 10,
batch_size = 32,
validation_data=(X_test, y_test)
)
estimator.fit( X_train, y_train )
出力
Epoch 8/10 Epoch 8/10 961/961 [==============================] - 0s - loss: 0.2449 - acc: 0.0114 - val_loss: 0.3433 - val_acc: 0.0187 Epoch 9/10 961/961 [==============================] - 0s - loss: 0.2063 - acc: 0.0135 - val_loss: 0.4258 - val_acc: 0.0125 Epoch 10/10 961/961 [==============================] - 0s - loss: 0.2354 - acc: 0.0114 - val_loss: 0.2858 - val_acc: 0.0187
y_pred = estimator.predict(X_test)
rms = (np.mean((y_test - y_pred) ** 2)) ** 0.5
#s = np.std(y_test - y_pred)
print("Neural Network RMS", rms)
Neural Network RMS 0.53456464492
可視化
import matplotlib.pyplot as plt
plt.figure()
plt.scatter(y_train, estimator.predict(X_train), label = 'Train', c = 'blue')
plt.title('Neural Network Predictor')
plt.xlabel('Measured Solubility')
plt.ylabel('Predicted Solubility')
plt.scatter(y_test, estimator.predict(X_test), c = 'lightgreen', label = 'Test', alpha = 0.8)
plt.legend(loc = 4)
plt.show()
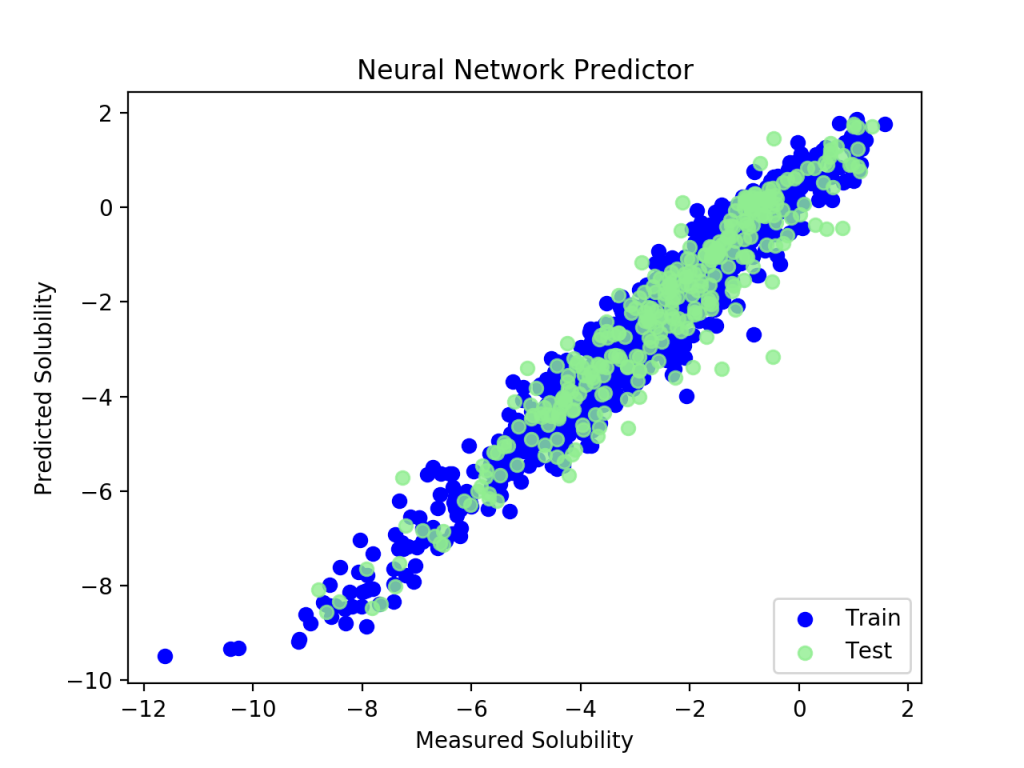
(了)
<<付録>> 部分最小二乗回帰 (PLSR)
参考サイト: scikit-learn: Partial Least Squares
ケモメトリックス (化学計量学) 分野で使われる部分最小二乗回帰 (Partial least squares regression) ではどうでしょうか.
import numpy as np
from sklearn.preprocessing import StandardScaler
from sklearn import model_selection
from sklearn.metrics import mean_squared_error
from sklearn.metrics import r2_score
from sklearn.cross_decomposition import PLSRegression
import sklearn
print("sklearn ver.", sklearn.__version__)
print("numpy ver.", np.__version__)
#保存済みファイルを読み込む
X = np.load("X_2d.npy")
Y = np.load("Y_2d.npy")
#訓練セットとテストセットにランダムで分割
X_train, X_test, y_train, y_test = model_selection.train_test_split(X,
Y, test_size = 0.25, random_state = 42)
#溶解度の分散をよく説明する因子を計算し (主成分分析はデータをよく説明する因子を計算する),15番目までの因子を使って回帰分析を行う.
pls2 = PLSRegression(n_components = 15, scale = True)
pls2.fit(X_train, y_train)
pred_train = pls2.predict(X_train)
pred_test = pls2.predict(X_test)
rms = (np.mean((y_test - pred_test)**2))**0.5
#s = np.std(y_test - y_pred)
print("PLS regression RMS", rms)
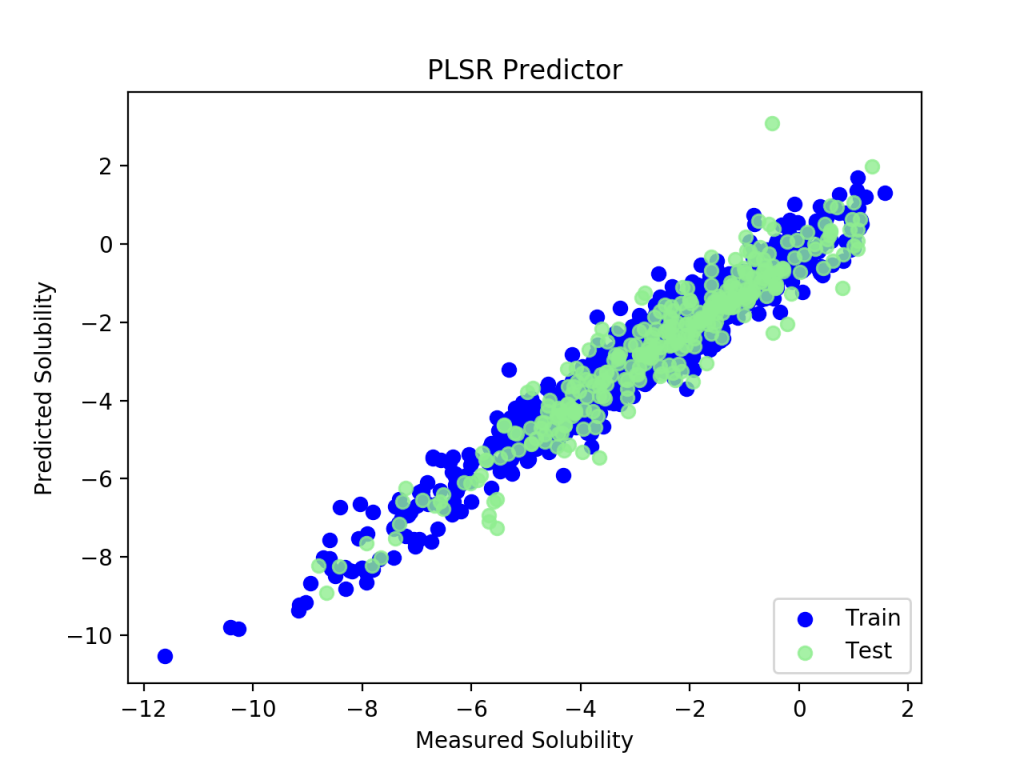
#可視化
import pylab as plt
plt.figure()
plt.scatter(y_train, pred_train, label = 'Train', c = 'blue')
plt.title('PLSR Predictor')
plt.xlabel('Measured Solubility')
plt.ylabel('Predicted Solubility')
plt.scatter(y_test, pred_test, c = 'lightgreen', label = 'Test', alpha = 0.8)
plt.legend(loc = 4)
plt.show()
PLS regression RMS 2.82767316393,でした.きれいに回帰できていました.計算は一瞬で終わりました.
RCY様、始めまして、化学原料コストダウン研究所所長の山本恒雄(75歳)と申します。ここ半年間機械顎数を始めております。この報文が素晴らしいので是非ともトライして、参考にさせていただきたいと思っております。しかしあいにく、前段の準備が上手く咀嚼できておりません。私のPC(下記の環境下)でやるべきアクション(多分、Anaconda prompt上での処理で対応可能?)をご教示いただけませんでしょうか?
・PCはWIN10
・Pythonはインストール済み
・anaconda一式、Note Bookもインストール済み
・RDKitとmordredは準備済み
・openbabelはインストール済み
・KerasとTensorFlowはlistにありません(これだけ準備すれば良いのかも?)
では、ご返事の程、よろしくお願い申し上げます。
山本 様
この記事の内容は古いため、より新しいものを参考にされることをお勧めします。
Anacondaが導入済みであれば、KerasやTensorflowは、condaコマンドからインストールできるのではないでしょうか。Windowsは使用していないため詳細は分かりかねます。