Contents
AlphaFold2
Alphabet Inc. 傘下のDeepMindが開発し、圧倒的な性能を見せるタンパク質立体構造予測プログラムAlphaFold2が公開されSNSでも大盛り上がりでした。
主な生物の全タンパク質の予測立体構造データベースも公開されました。
ローカル環境へのインストール(AlphaFold (ver.2) インストール@Ag_smith)はディスク容量など大変そうですが、Google Colaboratoryから有志が公開した簡易版が利用可能でデータベースにない配列、オリゴマー、ヘテロ複合体などの構造予測ができます。
Protein kinase Cファミリーはどうなっているか
そこで、protein kinase Cファミリー、C1ドメインを含むタンパク質の予測構造を見てみるわけです。
- PKCα (human) https://alphafold.ebi.ac.uk/entry/P17252
- PKCβ (human) https://alphafold.ebi.ac.uk/entry/P05771
- PKCγ (human) https://alphafold.ebi.ac.uk/entry/P05129
- PKCδ (human) https://alphafold.ebi.ac.uk/entry/Q05655
- PKCε (human) https://alphafold.ebi.ac.uk/entry/Q02156
- PKCη (human) https://alphafold.ebi.ac.uk/entry/P24723
- PKCθ (human) https://alphafold.ebi.ac.uk/entry/Q04759
- PKCζ (human) https://alphafold.ebi.ac.uk/entry/Q05513
- PKCι (human) https://alphafold.ebi.ac.uk/entry/P41743
ドメイン単位ではX線結晶構造解析で構造がいくつも解かれていて、相同性も高いので、テンプレートを使ったホモロジーモデリングが有効で、AlphaFold2でも実際そうしているでしょう。
Predicted aligned errorが低い領域で示される信頼性の高いドメイン間配置を探すと興味深かったのは、
- 連続したC1AドメインとC1Bドメインがどれも同じような配置で相互作用している、かつそのドメイン間相互作用が信頼性が高いとプログラムが評価していた点(predicted aligned errorをプロットした図上で緑色のクロスピークが出る; Google Colaboratory版では青色)。
- PKCγとPKCθでは、偽基質 (pseudosubstrate) 配列(α/β/γではRKGALR; δ/θ, RRGAIK; ε, RQGAVR; η, RQRAMR; ζ/ι, RRGARR) (Gong, et al., 2016) が触媒ドメインにきちんと蓋をしている構造が予測されている。PKCθではドメイン間配置の信頼性を示すpredicted aligned errorの値もちゃんと低い(緑色)。
- PKCγ, ε, ηではC1A+C1BとC2/C2-likeドメイン間の相互作用が信頼性が高い。
- PKCζ, ιのC1ドメインは間にβストランドを1本挟んでN末端のPHドメインと逆平行βシートを形成している。
しかし実験的に解かれたPKCβのほぼ全長構造中のC1Bドメインと触媒ドメインの配置が真だとして、そのC1BドメインにAlphaFold2が予測したようにC1Aを結合させると、今度は偽基質配列が触媒ドメインの活性部位に届かなくなり、矛盾が生じます。やはり、PKCβの結晶構造で電子密度が見えていないことからも分かる通り、C1Aドメインはかなりふらふらした位置にあり、不活性型酵素ではC1Bドメインと強固に相互作用していないと考えられます。ただし、C1Bドメインが触媒ドメインから外れて酵素が活性型になった場合は、AlphaFold2が予測したようにヘテロ二量体のようになる可能性もあります。
C1AとC1Bドメインの相互作用
C1ドメインについては、どれも(ドメインを顔に例えると)C1A、C1Bドメインとも上下は同じ向きに揃っていて、C1Bドメインが鼻をC1Aドメインの右頬に押し付けているような配置です。C1AドメインとC1Bドメインの間は約20残基のリンカー部位があります。
これが約70残基離れているPKDではC1AとC1Bドメイン間のpredicted aligned errorは高く、信頼性がないとされています。
- PKD1 (human) https://alphafold.ebi.ac.uk/entry/Q15139
リンカーの存在や長さが影響しているか調べようと、Google Colaboratory版を使って複合体構造の予測を行いました(ドメイン間にアミノ酸記号U(Unknown)× >32個を挿入する)。こういうことが簡単にできてしまうのが便利です。
PKCαの場合はリンカーがあってもなくても上記の複合体構造が予測としてでてきます。
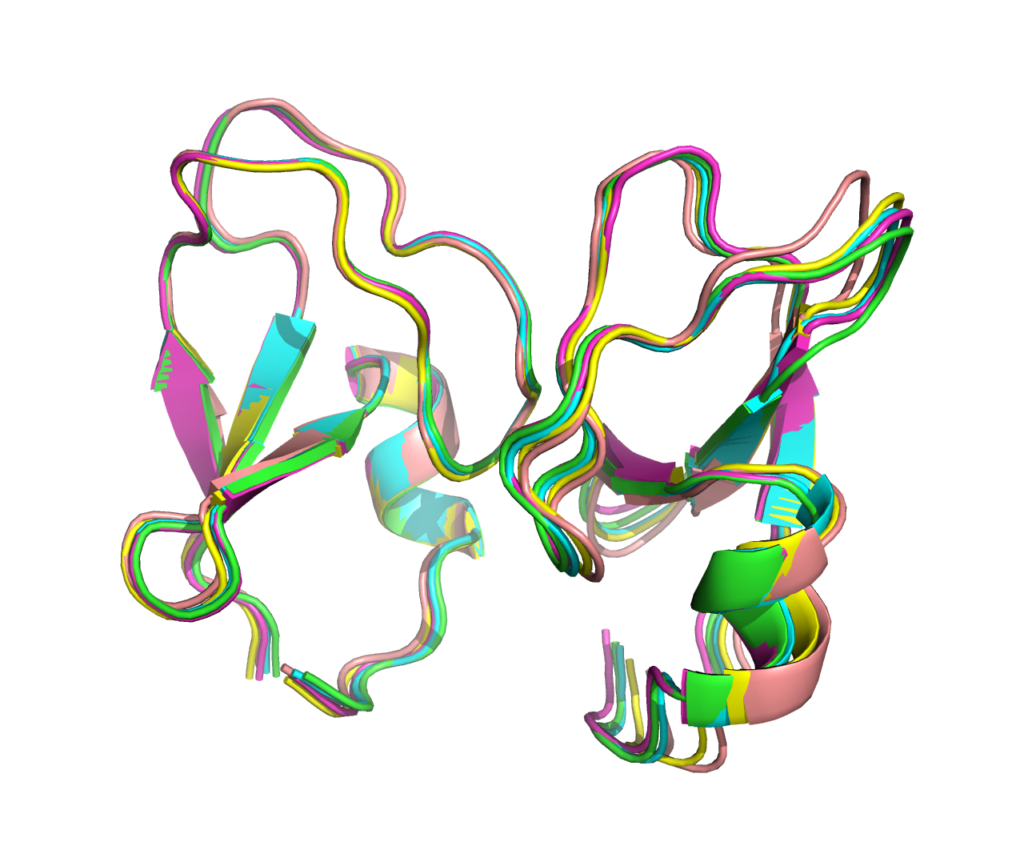
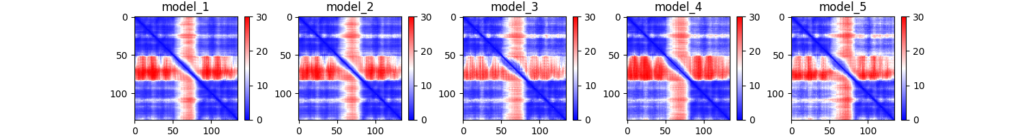
ドメインの配置の信頼性も高いです(対角線上の青い四角のブロックが各ドメインのフォールディングの信頼性、右上/左下の青色ブロックがドメインの配置の信頼性が高いことを示す。赤い部分はUnknown linkerの部分)。
対して、PKD1の場合は一定した複合体構造は予測されず、ドメインの配置の信頼性も低い結果でした。
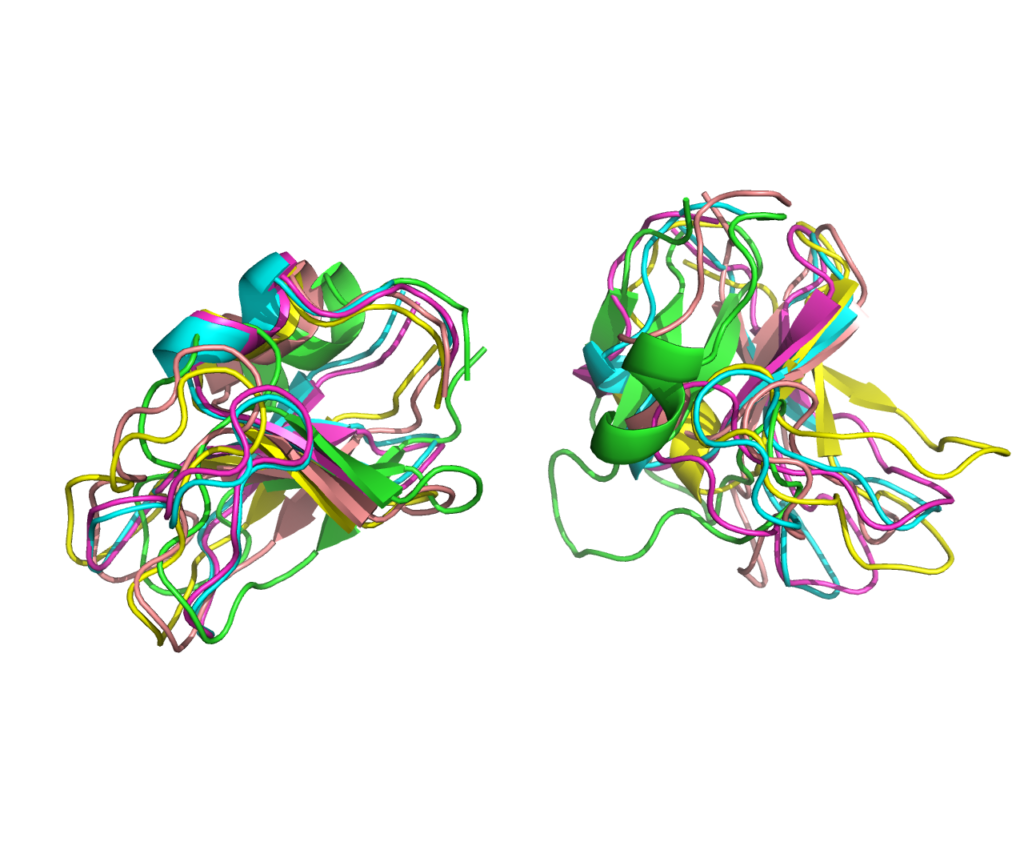
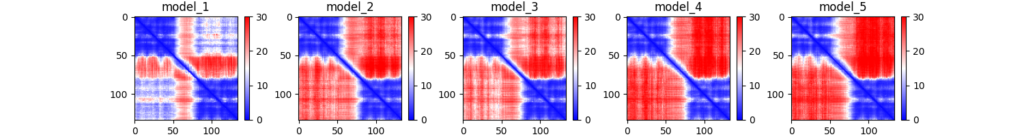
AlphaFold2の中身を理解していないので、どういった根拠で信頼性の高い低いを判断しているのかは分かりません。同様に複数(2つまたは3つ)のC1ドメインを持つdiacylglycerol kinase (DGK) ファミリーではC1ドメイン間でもC1ドメイン–他ドメイン間でも配置の信頼性が高い予測構造が出ています。
出て来た立体構造のエネルギー最小化をする場合、自分で亜鉛イオンを挿入して、以前書いた方法でAMBERかGROMACSを使って実行できます。
二量体型C1ドメインリガンド
PKCには2つのC1ドメインが並んで存在しますが、天然からは二量体型C1ドメインリガンド (blastmycetin A) の単離が報告されています。
-
Hagiwara, N.; Irie, K.; Funaki, A.; Hayashi, H.; Arai, M.; Koshimizu, K. Structure and Tumor-promoting Activity of New Teleocidin-related Metabolites (Blastmycetins) from Streptoverticillium blastmyceticum. Agric. Biol. Chem. 1988, 52, 641–648. DOI: 10.1271/bbb1961.52.641
- Wender, P. A.; Kehler, M. F. T.; Wright, D. L.; Irie, K. Mapping Phorbol Ester Binding Domains of Protein Kinase C (PKC): The Design, Synthesis and Biological Activity of Novel Phorbol Ester Dimers. Synthesis 1999, S1, 1401–1406. DOI: 10.1055/s-1999-3656
- Sridhar, J.; Wei, Z.; Nowak, I.; Lewin, N. E.; Ayres, J. A.; Pearce, L. V.; Blumberg, P. M.; Kozikowski, A. P. New Bivalent PKC Ligands Linked by a Carbon Spacer: Enhancement in Binding Affinity. J. Med. Chem. 2003, 46, 4196– 4204. DOI: 10.1021/jm0302041
- Ohashi, N.; Kobayashi, R.; Nomura, W.; Kobayakawa, T.; Czikora, A.; Herold, B. K.; Lewin, N. E.; Blumberg, P. M.; Tamamura, H. Synthesis and Evaluation of Dimeric Derivatives of Diacylglycerol–Lactones as Protein Kinase C Ligands. Bioconjugate Chem. 2017, 28, 2135–2144. DOI: 10.1021/acs.bioconjchem.7b00299
これらの最適なリンカー長を今回の予測構造から合理的に説明できるのでしょうか。上記の2番目の論文(Sridhar et al., 2003)のリガンド単量体をPKCα C1A-C1Bドメインの予測複合体構造に配置して距離を測ってみました。
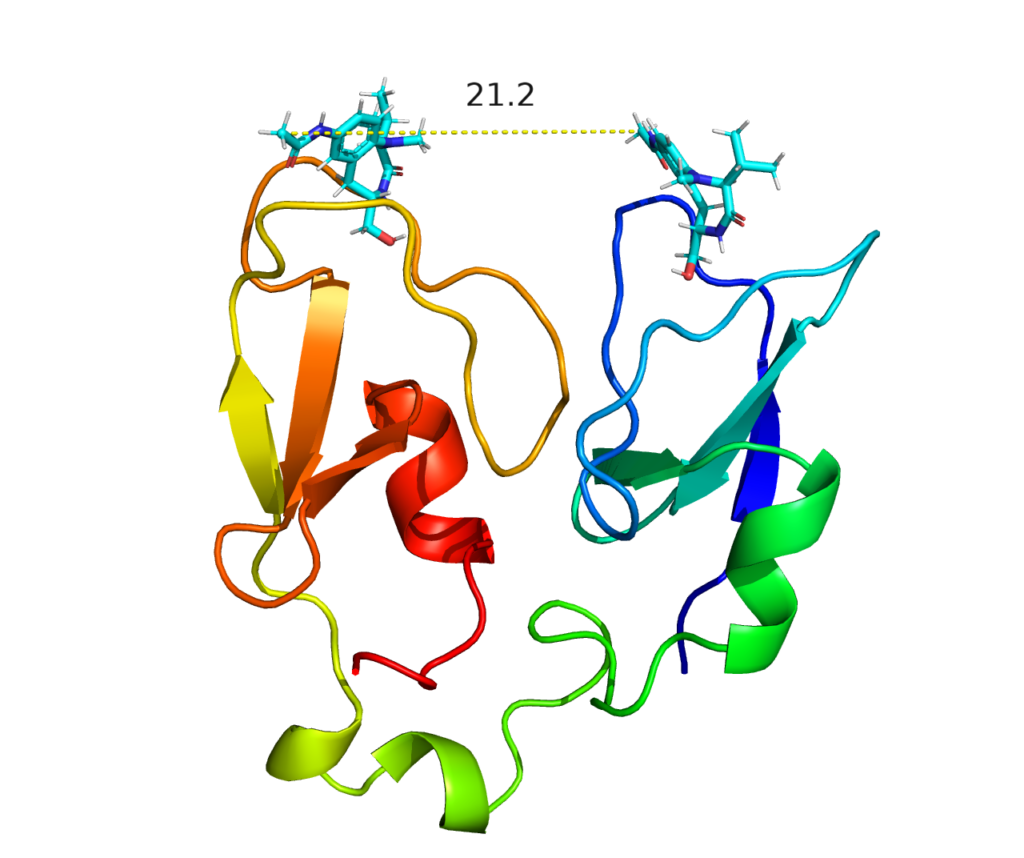
アルキル直鎖でつながれていた炭素間の距離は直線でも21 Åあって、論文で最適だった炭素数14では届きそうもありません(つないでみたら炭素が23個必要でした)。
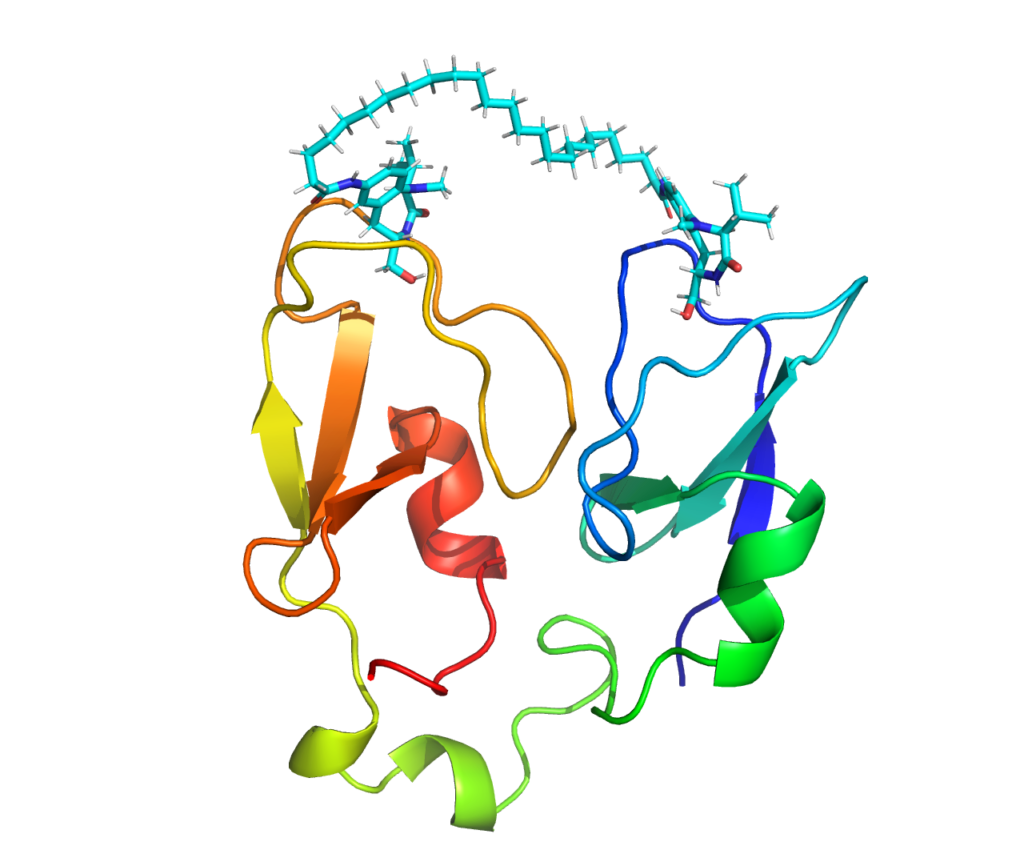
設計の意図通りにダイマーが2つのドメインに結合するためには、ドメイン側がAlphaFold2の予測構造とは別の配置を取る必要があることが分かりました。
メモ:
- PKCβIIのほぼ全長結晶構造(https://www.rcsb.org/structure/3pfq)で見られたC1Bドメインのリガンド結合cleftとキナーゼドメインとの相互作用はAlphaFold2では再現されていなかった。
- PKCβIIの結晶構造ではC1Aドメインの電子密度は見えていないので、C1AとC1Bドメインの複合体構造はそれほど強いものではない。
- PKCの基質モチーフ配列はRXXS/TXRX(Xは任意のアミノ酸)(Pearson & Kemp, 1991)。
参考文献
- Gong, J.; Holwinski, R. J.; Van Eyk, J. E.; Steinberg, S. F. A novel phosphorylation site at Ser130 adjacent to the pseudosubstrate domain contributes to the activation of protein kinase C-δ. Biochem J. 2016, 473, 311–320. DOI: 10.1042/BJ20150812. PMCID: PMC5022567.
- Pearson, R. B.; Kemp, B. E. Protein kinase phosphorylation site sequences and consensus specificity motifs: Tabulations. Methods Enzymol. 1991, 200, 62–81. DOI: 10.1016/0076-6879(91)00127-I
その他のC1ドメイン含有タンパク質へのリンク
DAG結合能を持つもの
DAG結合能を持たないもの
(了)